Complémentarité entre l'ordinateur et le cerveau dans le domaine de la biologie
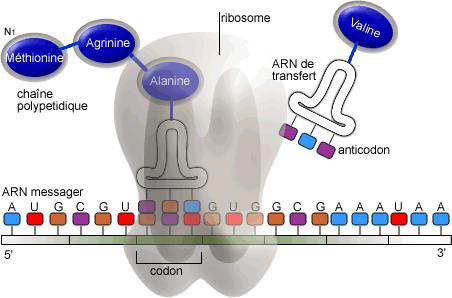
Une chaîne polypeptidique ou protéine est une suite d'acides aminés formés par les ribosomes. Elles ont de multiples fonctions, puisque ces chaînes sont entre autres utilisées dans notre corps comme par exemple, l'hémoglobine qui est composée de 4 chaînes polypeptidiques similaires deux à deux, et qui transporte l'oxygène dans notre sang. Elles sont aussi utiles partout dans la nature et l'exemple le plus flagrant est celui de la photosynthèse. En effet celle ci est faite par un Photonsysteme I, assemblage complexe de protéine qui capture chacune d'elles un photon ( énergie lumineuse ) et le transforme en énergie ( ici énergie chimique ). Cependant, contrairement à ce que l'on pourrait penser à première vue, ces chaînes ne sont pas planes et possèdent une forme en 3 dimensions. Cette molécule tendra à prendre une forme spécifique, à cause de différentes intéractions, liaisons et autres phénomènes. On dit que la molécule se plie. Cependant, il nous est encore impossible, et cela, malgré de nombreuses recherches, de prévoir la façon dont une molécule va se plier et grâce à quelle intéractions. C'est-à-dire que n'avons pas de lois qui préciserait qu'un enchaînement XYZ se plierait de cette façon, car ceci intéragit avec cet autre élément. Il est donc néscessaire de mener des recherches pour chaque protéines et pour chaque pliage. Nous savons cependant que la forme (en 3D) que prendra la molécule interférere sur ses capacités à interagir avec son environnement, voir peux la rendre complètement inéfficace . Ainsi, certaines molécules qui se sont mal pliées peuvent être responsable de maladies héréditaire comme la fibrose kystique et l'emphysème héréditaire, mais aussi d'autres maladies tristement célèbres : comme celle d'Alzheimer pour certains cas et le cancer qui n'est dangereux que quand la protéine P53 est défaillante, ce qui peut être dû à un mauvais pliage.
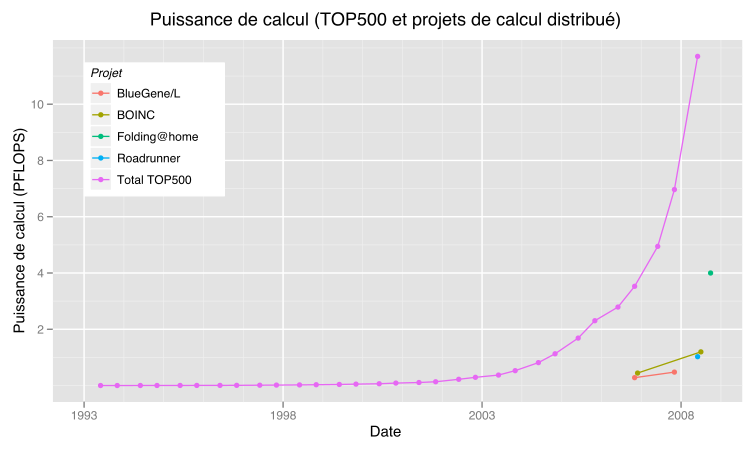
Pour chercher la façon dont se plie les protéines, et par défaut pour trouver comment un maximum d'entre elles se plie, l'université de Stanford à décidé de mettre en place à travers le monde un réseau d'ordinateurs qui prêterait la puissance que chaque ordinateur de ce réseau n'utiliserait pas, grâce au programme folding@Home chaque ordinateur reçoit un petit morceau de protéines à plier nommé « Unité de travail » et teste toutes les possibilités jusqu'a obtenir un schéma jugé convenable par l'algorithme développé et gardé secret par Stanford. L'intérêt de ce projet réside dans le fait de permettre à n'importe qui, qui y serait participant et qui le veut de prêter la puissance de son CPU sans pour autant que cela ne le gène, puisque le programme s'adapte automatiquement à la puissance requise par les autres programmes de votre ordinateur (on dit qu'il lui est alloué une priorité minimale sur les tâches). De nombreuses personnes rejoignirent ce réseau, dont de nombreux grands acteurs comme Google qui mettra l'action en avant grâce à son célèbre moteur de recherche ou Sony, qui intégrera ce programme à sa console de jeu PlayStation 3. La puissance de calcul mise en commun est actuellement de 4 petaflops soit 4*10^15 calculs de nombres à virgule flottante par secondes, c'est-à-dire deux fois plus puissante que le supercalculateur chinois Tianhe-IA et 3 fois plus puissante que Jaguar, le supercalculateur américain. Il est possible de voir les résultats que cette méthode a apportés au domaine de la science sur le site internet suivant : http://folding.stanford.edu/French/Papers . Les résultats sont donc conscéquent.
Toute fois, il ne faut pas oublier que cette méthode tente toutes les solutions possibles pour chaque pliage de chaque protéine. Cela peut prendre beaucoup de temps, en effet une unité de travail prend de quatre à mille heures soit presque 5 semaines de calculs. Ici, l'ordinateur n'utilise aucune logique. Beaucoup de gens qui regardaient l'ordinateur effectué (le programme disposant d'un système de modélisation des protéines qui pouvait être utilisé en tant qu'écran de veille) ces pliages s'étonnaient, du chemin logique pris par l'ordinateur pour résoudre un pliage quand la solution leur paraissait si évidente. En conséquence, l'université de Stanford sortit un autre logiciel dans un autre genre que folding@Home nommé Foldit. Ce programme se présente sous forme d'un jeu/puzzle, durant lequel l'humain résout le pliage de protéines munies de sa souris avec laquelle il peut appliquer des vecteurs sur chaque partie. Notre sens de la visualisation en 3 dimensions, inné, a permis d'obtenir de surprenants et positifs résultats. Les meilleurs « joueurs » sont capables de résoudre un pliage de molécule 100 fois plus rapidement qu'un ordinateur. Il leur est donc alloué préférentiellement des unités de travails qui prendraient normalement 1000 h pour un ordinateur.
Nous avons vu que foldit@home « forçait » le pliage des protéines en traitant toutes les possibilités de pliage, et en gardant à chaque fois la plus correcte des formes, et nous avons aussi vu un second logiciel, Foldit, qui proposait à des humains de plier des molécules sous la forme d'un jeu. Listons maintenant les avantages et inconvénients de chacune de ces deux méthodes :

Voyons maintenant comment ces avantages et inconvénients sont utilisés pour tirer le plus de profit.
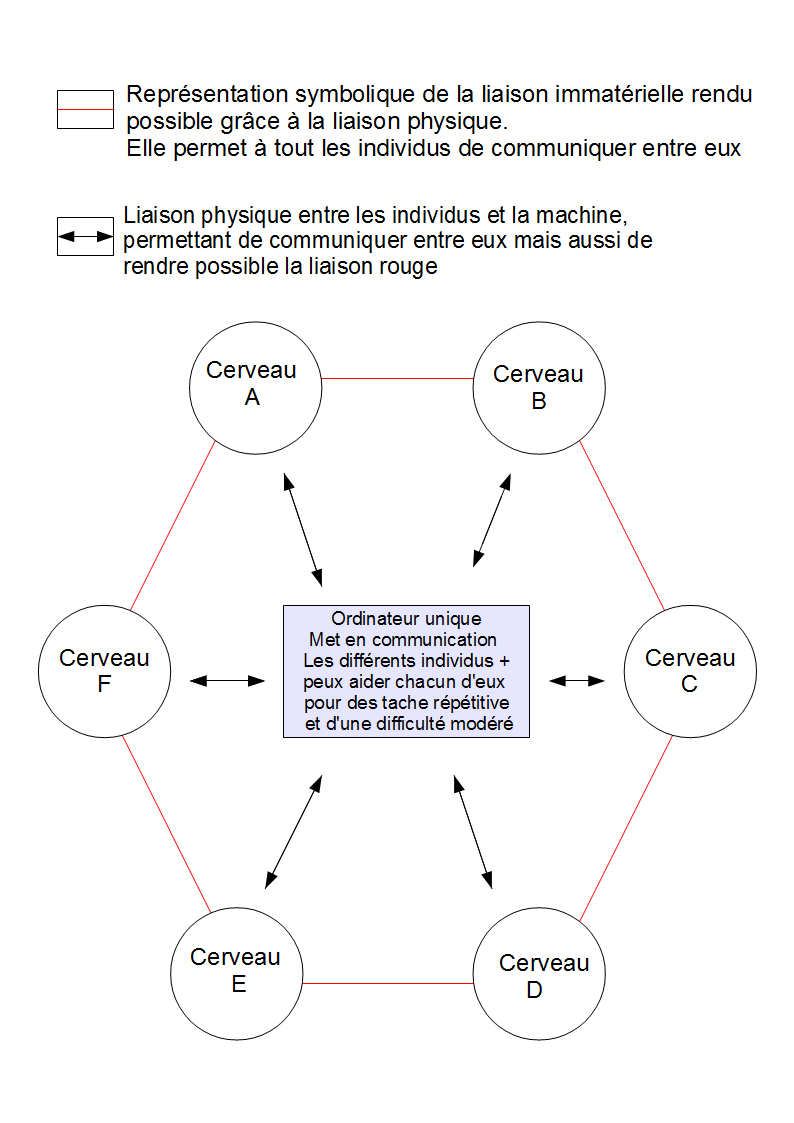
Il y a deux approches de travaux ordinateur-cerveau pour le même problème.
Dans le cas de Folding@Home :
L'ordinateur traite l'unité de travail, le renvoi au serveur mère de Stanford, qui lui même la transmettra à une équipe de chercheurs. Ces chercheurs analyseront les résultats, effectuerons différentes comparaisons et pourront ainsi en déduire les réactions chimiques qui ont amené la molécule à se plier de cette façon. Et c'est la compréhension de ce phénomène, et de comment arriver à cette forme finale, qui permet de développer des médicaments adaptés aux différentes anomalies ou maladies. L'ordinateur effectue donc un travail préliminaire, qui sera ensuite complété et approfondi par un chercheur, donc un cerveau humain. Encore une fois l'ordinateur effectue des travaux bruts et fastidieux dans cet exemple. Cependant, voyons maintenant une autre façon d'utiliser l'ordinateur dans la résolution de problème, à travers l'exemple de Foldit.
En premier lieu, l'ordinateur est le support du programme. Ce point peut paraitre anodin, mais il ne serait pas possible de jouer à plier des molécules via un jeu de société sous forme de plateau.
Tout comme Folding@home, il transmet vos résultats au serveur de Stanford qui effectuera les mêmes actions. Cependant, rappelons-nous qu'un cerveau humain possède un rendement pour ce genre de tâche 10 fois supérieur à celui de l'ordinateur c'est pourquoi le programme Foldit permet de mettre en commun des cerveaux à travers le monde. Ainsi nous nous retrouvons avec une architecture similaire à Folding@Home, et nous accédons à une nouvelle voie de travail : la mise en commun de capacité cérébrale. Et c'est en créant et en entretenant des liens entre les cerveaux des participants que l'ordinateur est de mise avec l'homme pour résoudre communément des problèmes. Car sans lui il ne serait pas imaginable de regrouper à toute heure du jour et de la nuit des joueurs avides. De plus, le joueur peux très bien se faire aider par l'ordinateur en lançant un des 2 outils mis à sa disposition: premièrement un outil qui réoriente certaine partie de la molécule de façons très légères, ce qui n'influe pas sur la direction et la forme globale de la protéine pliée, soit de lancer un outil qui tentera toutes les différentes possibilités de pliage, de la même façon que Folding@Home ce qui n'as donc ici qu'un intérêt limité.
L'expérience a confirmé la puissance de Foldit, comme le montre l'anecdote suivante :
Les chercheurs dans ce domaine étaient bloqués depuis plus de 10 par la complexité de la protéase rétrovirale du virus M-PMV (Mason-Pfizer monkey virus). Il leur était impossible de la plier et donc de trouver sa forme en 3 dimensions. Ils décidèrent de poster cette protéine sur foldit et seulement 3 semaines après les utilisateurs de Foldit avaient trouvé sa forme pliée, ce qui valu une parution sur le magazine Nature Structural & Molecular Biology. Il ne reste plus qu'au biologiste de trouver une molécule capable d'arrêter le M-PMV, ce qui reste tout de même une tâche considérable.

Conclusion
Dans cette partie nous avons vu une application du travail ordinateur-cerveau, présenté sous deux formes et deux approches différentes pour résoudre le même problème. Premièrement folding@home qui utilise l'ordinateur pour faire un travail préalable et secondairement foldit qui l'utilise comme moyens de communication et de synchronisation intercérébrale qui permet donc un traitement des données communs (cf schéma), mais aussi comme assistance pour des tâches plus difficiles à l'humain. C'est à travers ces deux procédés que la recherche sur le pliage des protéines a pu grandement avancé.